【NGS Introduction Series-Bacterial Whole Genome Sequencing Analysis and Applications】
在介紹過細菌WGS是如何進行的之後,今天來告訴大家實驗完成後得到的數據後續要如何處理呢?
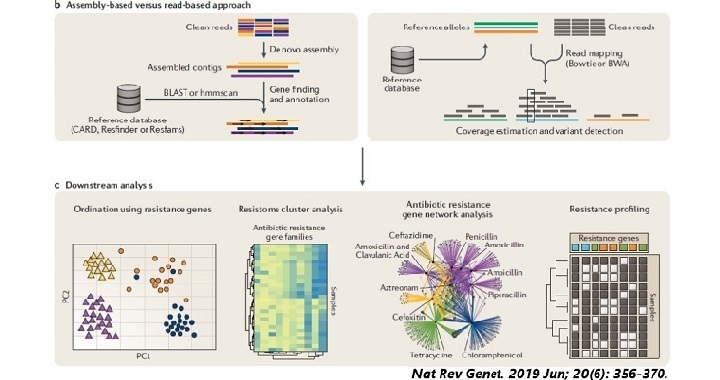
- 沒有參考序列(reference genome)時會使用De-novo assembly方法
- 有參考序列會使用Reference Mapping方法
1. De-novo assembly組裝
在沒有參考序列的情況下,僅使用測序的序列片段的資訊來組裝的方法。
利用將reads的末端序列相同的區域連結合併起來,形成contig
希望最後可以得到一條還原後的完整基因組序列(genome);
但是有些情況會使組裝遭遇困難,例如:定序錯誤、核酸多型性、定序偏差(sequencing bias)…….
這時候可以解決的方法為:
- 加深定序深度
- 組裝前先依據序列的品質分數(quality score)進行裁切(trim)
- 使用PCR free的方法製備樣品
2. Reference mapping
在有參考序列的情況下,將測序片段比對(mapping)至參考序列上,以取得分析結果。
Reference mapping的方式通常較為快速且對計算的要求較低,
對於參考序列的資料庫選擇非常重要,
分析結果的品質通常透過3個量化指標來評估:
- Mapping rate: Mapped reads/Total reads
- Genome coverage: Mapped genome/Total genome
- Depth: 平均深度及單鹼基的深度分布
近年來隨著NGS技術持續提升和生物資訊分析技術不斷進步,加上定序成本的降低,
細菌WGS在流行病學及公衛領域的使用逐漸廣泛,
如:抗藥性基因檢測、菌株基因分型、聚集事件調查、藥物疫苗開發…等
另外,在食品微生物的檢測方面,隨著大量微生物定序資料的快速累積,
能夠更有效率去追溯食品汙染源及傳播途徑,
甚至能藉此改善食品的製造或運輸程序以增進食品品質與安全,
NGS在疾病預防控制與民生食安上已成為不可或缺的工具了!
Next Generation Sequencing (NGS) Sequencing Service
1. Nanopore and Illumina offer a complete range of sequencing platforms for long and short sequences.
2. An excellent choice for high efficiency and quality.
3. Professional sample quality control processes and library preparation.
4. Professional bioinformatics analysis and service consultation.