掌握引子設計的要點,能幫助您有效並精準地進行PCR實驗與定序分析,一般PCR與定序引子設計時可注意以下幾點:
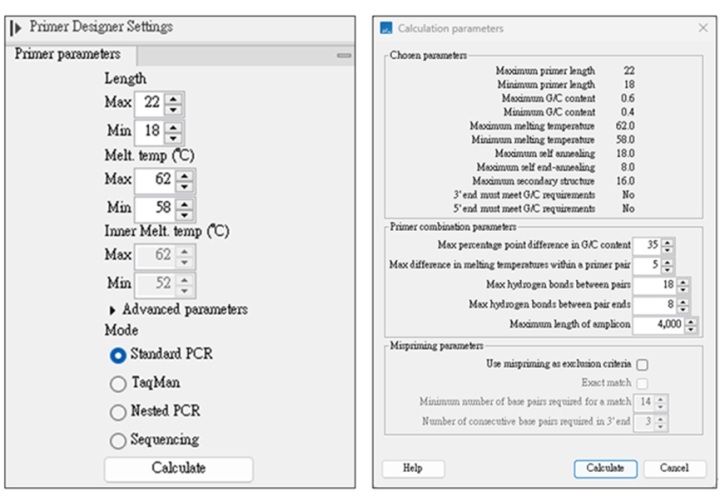
1. 定序與PCR引子通常建議介於18-25 mers,GC%介於30-80%
2. 引子必須檢查是否與目標序列只有一個binding site,若有二個(含)以上,定序和PCR時會呈現多種序列。
3. 為確保引子專一性,於3’端至少要有10個nucleotide與目標序列完全互補,且3’端避免2 組以上 G+C(GCGC),因為可能黏合上序列中GC rich區域。另外,定序引子盡量避免為複合序列。
4. 避免連續的鹼基出現,連續鹼基在引子合成時容易產生合成不完全的現象,尤其連續 4 個以上的 G 出現,還會大幅提高引子的Tm值。
5. 定序使用的引子建議Tm值介於50-58 ℃,若您的PCR引子Tm值較高或更低,可能會不適用於定序反應,因為定序時的PCR反應之annealing溫度為50℃。
6. 3’端第一個 nucleotide 不為A,因3’端A於錯配(mismatch)發生時,polymerase仍有較大機率仍會進行extention。
7. 引子自身及引子之間若有連續4個鹼基互補,或含有限制酶切位,易造成self annealing。引子自身互補會形成Hairpin的二級結構,而引子間互補會形成primer dimer,皆不利於PCR與定序反應。
8. 引子的mismatch若集中在5’端,仍有能會與目標序列的其他位置黏合,需注意以免PCR或定序時會產生多種序列。
源資國際生物科技提供客製化的諮詢與高效率的引子合成服務,若您有任何關於PCR與定序引子的疑難雜症,歡迎請您與我們聯繫。
#詳盡的售後服務諮詢
#專屬的數據管理系統
#迅速的發送結果報告
#專業的生物資訊分析
#源資生技
#TriIBiotech
#引子設計
Next Generation Sequencing (NGS) Sequencing Service
1. Nanopore and Illumina offer a complete range of sequencing platforms for long and short sequences.
2. An excellent choice for high efficiency and quality.
3. Professional sample quality control processes and library preparation.
4. Professional bioinformatics analysis and service consultation.