16S metagenome目前常用來進行環境微生物族群分析的方法,藉由微生物體內的高度保留16S rRNA基因的序列來進行研究,以便能夠以宏觀的角度進行探討,研究環境中菌相的變化。
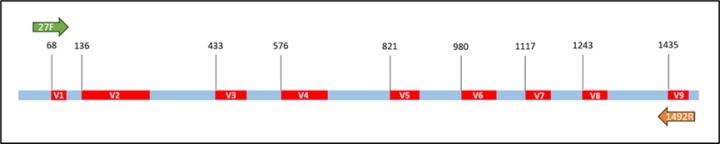
在16S rRNA基因上除了含有高度保留的區域之外,同時也存在著高度變異的區域 (如下圖),透過PCR的方式針對此區域進行放大 (常見區域為V3-V4區域),結合次世代定序的輔助進行定序。所獲得的數據依照原本PCR放大的片段大小進行處理,如果片段較大的話需要先進行Merge的步驟;如果片段較小的話可以直接使用單邊的序列進行後續分析。而依照不同的分析方法將相似的序列進行整合,獲得16S rDNA的Operational taxonomic unit (OTU)或是Amplicon sequence variants (ASV)等maker gene序列。此方式由於只著重在特定區域,此辨識的解析度會受到部分限制。近年來由於三代定序儀的現世,定序全長的16S rRNA基因日漸盛行,相較於前的特定區域的分析方式,此序列可以直接進行物種比對,鑑定的解析度也相較提高許多。
為了鑑定不同的微生物,16S的定序資料將會針對已知的微生物資料庫進行比對,透過比對到的資料庫訊息,進行界、門、綱、目、科、屬及種等不同層次的分類學分析。目前常使用的資料庫有SILVA、RDP及Greengenes等。如此將鑑別出來的物種進行比較,進而找出其組別之間存在的差異性,以了解其對環境的影響。
#詳盡的售後服務諮詢
#專屬的數據管理系統
#迅速的發送結果報告
#專業的生物資訊分析
#源資生技
#TriIBiotech
#16smetagenome
Next Generation Sequencing (NGS) Sequencing Service
1. Nanopore and Illumina offer a complete range of sequencing platforms for long and short sequences.
2. An excellent choice for high efficiency and quality.
3. Professional sample quality control processes and library preparation.
4. Professional bioinformatics analysis and service consultation.